Plateforme Ariadne : Criblage à Haut débit et Haut contenu
Plateformes Lilloises en Biologie & Santé – PLBS – UMS 2014 – US 41

Présentation
ARIADNE regroupe deux plateformes, ARIADNE-Criblage et ARIADNE-ADME, travaillant de manière indépendante mais complémentaire. Leur objectif est de découvrir des petites molécules chimiques utiles en thérapeutique ou pour explorer le vivant, et de caractériser leurs propriétés ADME. ARIADNE-Criblage regroupe un ensemble d’expertises et d’équipements dédiés au criblage de perturbateurs biologiques tels que des petites molécules chimiques ou des librairies de siRNA. ARIADNE-ADME permet quant à elle de quantifier les paramètres qui caractérisent la capacité des molécules à être Absorbées, Distribuées, Métabolisées et Eliminées in vivo (ADME).
ARIADNE-Criblage est constituée de trois plateaux. Le plateau HTS permet de miniaturiser des essais pharmacologiques cellulaires ou non, et de cribler à haut débit des librairies chimiques. Il permet d’évaluer à moyen débit l’activité de séries chimiques en cours d’optimisation et d’explorer leur mécanisme d’action. Les plateaux pour le High Content Screening HCS-L2 et HCS-L3 permettent de réaliser à haut débit et à haut contenu des criblages par microscopie quantitative dans un environnement de sécurité L2 ou L3.
Savoir-faire et services proposés :
La plateforme regroupe un ensemble d’expertise et d’équipements dédiés au criblage de petites molécules chimiques ou des librairies de siARN. La chaine d’équipements permet l’automatisation complète ou partielle des étapes de criblage, depuis la mise en plaque des perturbateurs provenant de chimiothèques ou de siRNA-thèque, jusqu’à la lecture de résultats. Le savoir-faire et la technicité du personnel permettent le développement de tests à haut-débit, miniaturisés (plaques 96, 384 puits) et automatisés pour le criblage in vitro, in bacterio ou in cellulo, sur cibles ou phénotypique. ARIADNE bénéficie de l’accès à des chimiothèques notamment la chimiothèque régionale d’environ 200.000 molécules chimiques, de banques de siARN ou de miARN. La plateforme possède également une expertise de marquage de cellules pour la domiciliation cellulaire, sur les approches CRISPR Cas9.
Les équipements sont localisés dans des laboratoires de sécurité biologique de niveau 1 à 3. Le plateau HTS en LNSB1 permet la gestion des librairies et le criblage à haut débit sur des lecteurs de plaques multimodes (avec des mesures en fluorescence, luminescence, absorbance …). Les plateaux HCS pour le criblage à haut contenu, installés grâce à l’Equipex ImagInEx BioMed et au soutien du Conseil Européen de la Recherche, reposent sur une technique de microscopie confocale automatisée et quantitative. Un premier système d’imagerie (IN cell Analyzer 6000) est entièrement automatisé en LNSB2. Un second système (IN cell Analyzer 6500) est disponible en LNSB3. Cette technologie dite “non destructrice” autorise le suivi dans le temps du même échantillon à la fois à court terme (phénomènes dynamiques) et à long terme (cinétique) et permet de mesurer plusieurs paramètres simultanément sur un même échantillon biologique.
De nombreux tests peuvent être mis en œuvre : tests enzymatiques, tests cellulaires phénotypiques ou sur cible, interactions protéine-protéine in vitro ou in cellulo, Thermal Shift Assay (TSA) et suivi d’engagement de cible… L’utilisation de la technologie acoustique Echo pour le transfert de nanogouttes de liquide permet de préparer les plaques de criblage et de tester des molécules en combinaison et en dose réponse.
Les demandes destinées à ARIADNE-Criblage peuvent être adressées par mail au responsable scientifique. Elles feront l’objet d’une discussion avec le porteur de projet puis d’une étude de faisabilité. Les projets répondant au cahier des charges seront acceptés dans l’ordre d’arrivée. Selon les cas, le personnel d’ARIADNE-Criblage peut former les utilisateurs pour un accès autonome mais payant à la plateforme, établir des devis pour un service, collaborer à une recherche de financement… Chaque projet est suivi par un membre du comité de pilotage et par le responsable technique.
Dans le cadre d’un accord partenarial, les projets peuvent être poursuivis avec les experts de Ariadne-Criblage ou leurs collaborateurs vers la caractérisation de cibles et l’optimisation des modulateurs : identification et engagement de cible, chimie médicinale, mesure des propriétés physicochimiques, études ADME et pharmacocinétique chez les rongeurs. Ces derniers travaux peuvent être réalisés avec la plateforme ARIADNE-ADME.
Actus
La plateforme organise une école thématique sur le criblage qui aura lieu dans les Hauts de France du 20 au 23 septembre 2022. Les inscriptions se font sur le site de l’école.
Les techniciens, ingénieurs et chercheurs d’ARIADNE se sont engagés dans la recherche d’un traitement contre la pandémie Covid-19 :
- Une molécule active in vitro sur l’infection par SARS-Cov-2 a été découverte par criblage sur cellules d’une librairie de 2000 médicaments sur le système HCS en LNSB3. Cette découverte est issue d’une collaboration avec les Dr Sandrine Belouzard et Jean Dubuisson du Centre d’Infection et d’Immunité de Lille (IPL) et les chercheurs de la société Apteeus.
- Le criblage HTS de l’intégralité de la librairie d’ARIADNE (env. 100 000 composés) a permis d’identifier plusieurs séries chimiques actives sur la protéase 3CLPro de SARS-Cov-2 à des concentrations sub-micromolaires. Ce projet est mené avec le Dr Julie Charton de l’unité U1177.
Membres
La plateforme Ariadne-criblage fait partie de l’UAR 2014-US41-Plateformes Lilloises en Biologie et Santé et de l’Infrastructure de Recherche Nationale ChemBioFrance.
La plateforme est labélisée et soutenue par le Gis IBiSA.
Son comité de pilotage associe des chercheurs du Centre d’infection et d’Immunité de Lille : Dr Priscille brodin et Dr Alain Baulard, ainsi que de l’Unité U1177 : Pr Benoît Déprez et Dr Florence Leroux.
Priscille BRODIN
DR Inserm, CIIL UMR9017 U1019
Benoit DEPREZ
Pr Univ Lille, U1177
Alain BAULARD
DR Inserm, CIIL UMR9017 U1019
Florence LEROUX
IR Inserm, U1177
Alexandre VANDEPUTTE
IE Univ Lille, CIIL UMR9017 U1019
Cyril COUTURIER
MCU Univ Lille, U1177
Valentin GUILLAUME
CE IPL, U1177
Julie DUMONT
AI Univ Lille, U1177
Nathalie DEBOOSERE
CE IPL, CIIL UMR 9017 U1019
Valérie LANDRY
CE IPL, U1177
Adrien HERLEDAN
IE Inserm, U1177
Sandrine WARENGHEM
Tech IPL, U1177
Publications
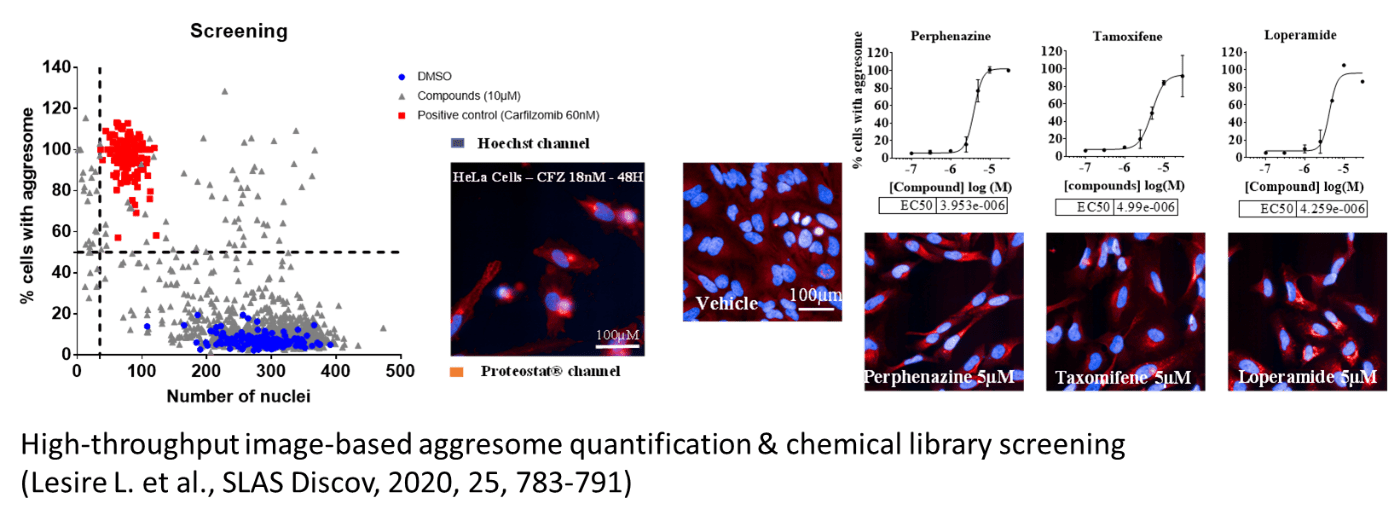
Lesire, L., Chaput, L., Cruz De Casas, P., Rousseau, F., Piveteau, C., Dumont, J., Pointu, D., Déprez, B., and Leroux, F. (2020).
High-Throughput Image-Based Aggresome Quantification.
SLAS Discov 25, 783-791.
Moraski, G.C., Deboosère, N., Marshall, K.L., Weaver, H.A., Vandeputte, A., Hastings, C., Woolhiser, L., Lenaerts, A.J., Brodin, P., and Miller, M.J. (2020).
Intracellular and in vivo evaluation of imidazo[2,1-b] thiazole-5-carboxamide anti-tuberculosis compounds.
PLoS One 15, e0227224.
Herledan, A., Andres, M., Lejeune-Dodge, A., Leroux, F., Biela, A., Piveteau, C., Warenghem, S., Couturier, C., Deprez, B., and Deprez-Poulain, R. (2020).
Drug Target Engagement Using Coupled Cellular Thermal Shift Assay-Acoustic Reverse-Phase Protein Array.
SLAS Discov 25, 207–214.
Blondiaux, N., Moune, M., Desroses, M., Frita, R., Flipo, M., Mathys, V., Soetaert, K., Kiass, M., Delorme, V., Djaout, K., Trebosc, V., Kemmer, C., Wintjens, R., Wohlkönig, A., Antoine, R., Huot, L., Hot, D., Coscolla, M., Feldmann, J., Gagneux, S., Locht, C., Brodin, P., Gitzinger, M., Déprez, B., Willand, N., and Baulard, A. R. (2017).
Reversion of antibiotic resistance in Mycobacterium tuberculosis by spiroisoxazoline SMARt-420.
Science 355, 1206–1211.